Nucleic Acids Research丨深圳先进院脑所陈宇团队等构建适用于“脑-肠轴”调控机制研究的质粒数据库
by 管理员 | 2024-10-29
肠道菌群是由大量微生物构成的复杂的微生态系统,参与了多种宿主生物调节过程,其与大脑之间的双向通讯机制构成了“脑-肠轴”,并与阿尔茨海默症、帕金森病和抑郁症等神经疾病的发生发展密切相关。近年来研究发现,质粒作为微生物的主要可移动遗传元件,不仅携带了抗性基因、毒力基因、代谢基因簇等多种功能元件,还可通过其多拷贝性和转移性影响肠道菌群功能及“脑-肠轴”通讯。尽管已有数据库对不同来源的质粒进行了归档,但这些数据库缺乏对质粒表型、基因型和功能的全面注释,且不同数据库间的数据隔离影响了质粒的联合分析。

文章上线截图
中国科学院深圳先进技术研究院(简称“深圳先进院”)脑认知与脑疾病研究所/深港脑科学创新研究院陈宇课题组和香港城市大学计算机系李帅成课题组于2024年10月23日在Nucleic Acids Research上在线发表了题为“PlasmidScope: A comprehensive plasmid database with rich annotations and online analytical tools”的研究论文,构建了一个包含全面注释、在线分析和交互可视化的质粒数据库,并探索了阿尔茨海默症肠道菌群中质粒的分布特征。
在本研究中,研究人员构建了PlasmidScope质粒数据库,收录并整理来自 10 个公共资源库的 852,600 个质粒,提供了其详尽的背景信息和注释结果,包括基因组完整性、拓扑结构、流动性、宿主来源、tRNA、tmRNA、信号肽、跨膜蛋白和 CRISPR/Cas 系统。同时,PlasmidScope 数据库对质粒的 25,231,059 个预测基因进行了多种功能注释,包括抗生素抗性、毒力因子及代谢基因簇等,并利用 ESMFold 软件预测对这些基因开展了蛋白三维结构预测。此外,PlasmidScope 还集成了在线分析模块和交互式可视化功能,支持不同数据间的交叉比较和查询,有助于研究人员对质粒进行深入研究(图1)。

图1. PlasmidScope数据库概览
随后,研究人员使用PlasmidScope解析了阿尔茨海默症患者肠道菌群中的质粒分布特征,并探索它们在“脑-肠轴”中可能发挥的作用。此前研究表明,脆弱拟杆菌在激活小胶质细胞和诱导阿尔茨海默症病变方面发挥着重要作用,因此,研究人员收集并分析了该细菌所携带的所有质粒。结果发现,脆弱拟杆菌共包含464种质粒,其中43种质粒不仅具完整环状基因组,而且具有可移动的特性。通过系统发育分析和功能预测发现,这43种质粒共携带1,726个基因,包括大量抗生素抗性基因和毒力基因(图2)。此外,质粒CP103137.1携带有两个与色氨酸合成相关的基因,为进一步研究脆弱拟杆菌与阿尔茨海默症之间的联系提供了重要线索(图2)。
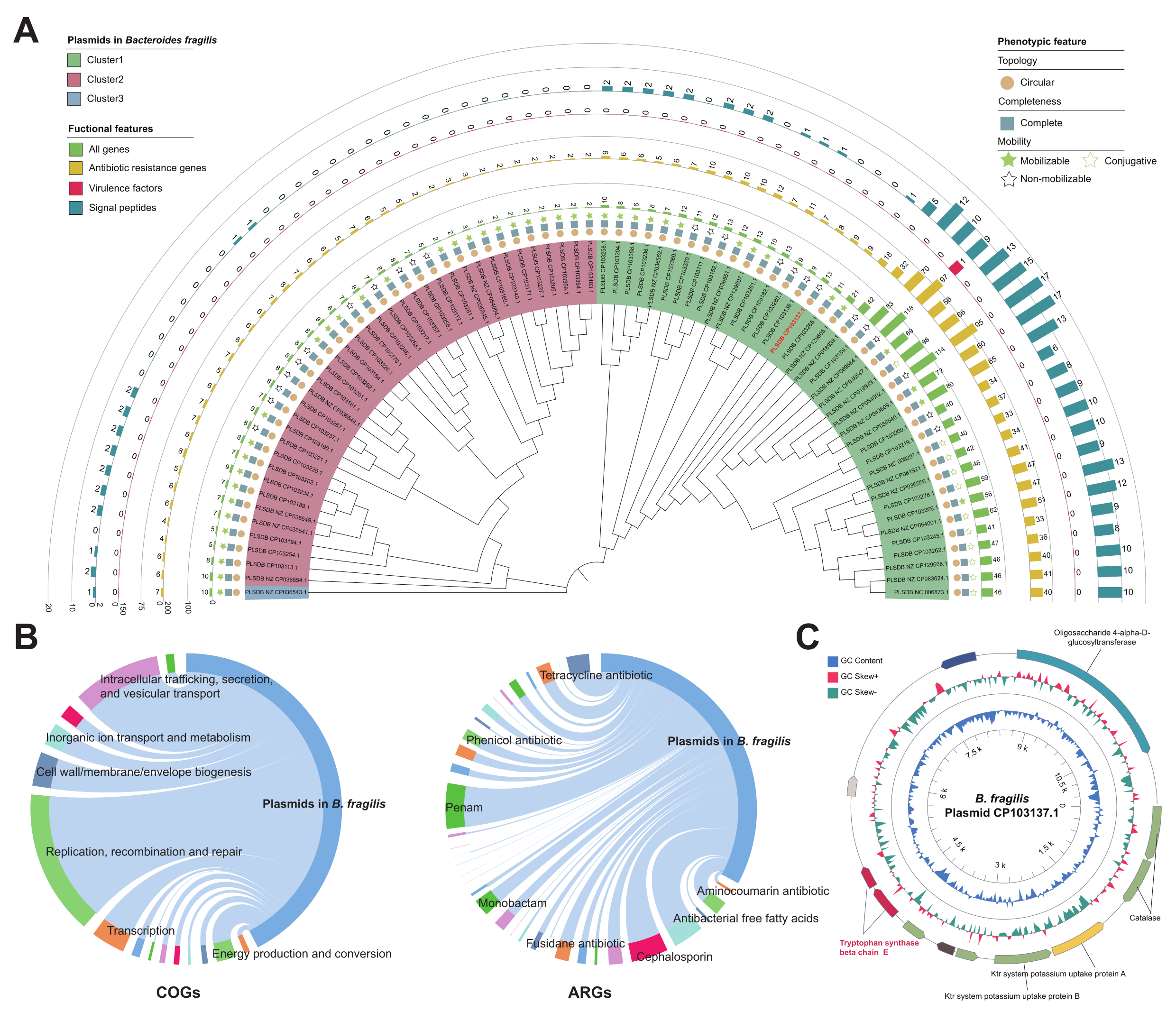
图2. PlasmidScope在研究阿尔茨海默症(AD)相关肠道菌群中质粒的应用
综上所述,本研究构建了一个质粒数据库PlasmidScope,收录了来自10个公共资源库的质粒数据,提供了全面的注释结果、交互式可视化和在线分析工具,有助于质粒结构和功能的解析,为进一步探索质粒在肠道菌群及“脑-肠轴”调控机制中的作用奠定了重要基础。
深圳先进院助理研究员李寅虎、研究助理陈姁桦和西北工业大学助理教授冯喜康为该论文共同第一作者,深圳先进院陈宇研究员和香港城市大学李帅成教授为该论文的共同通讯作者,深圳先进院为论文第一单位。最后,感谢国家自然科学基金、广东省自然科学基金等研究计划对本项研究的资助。

